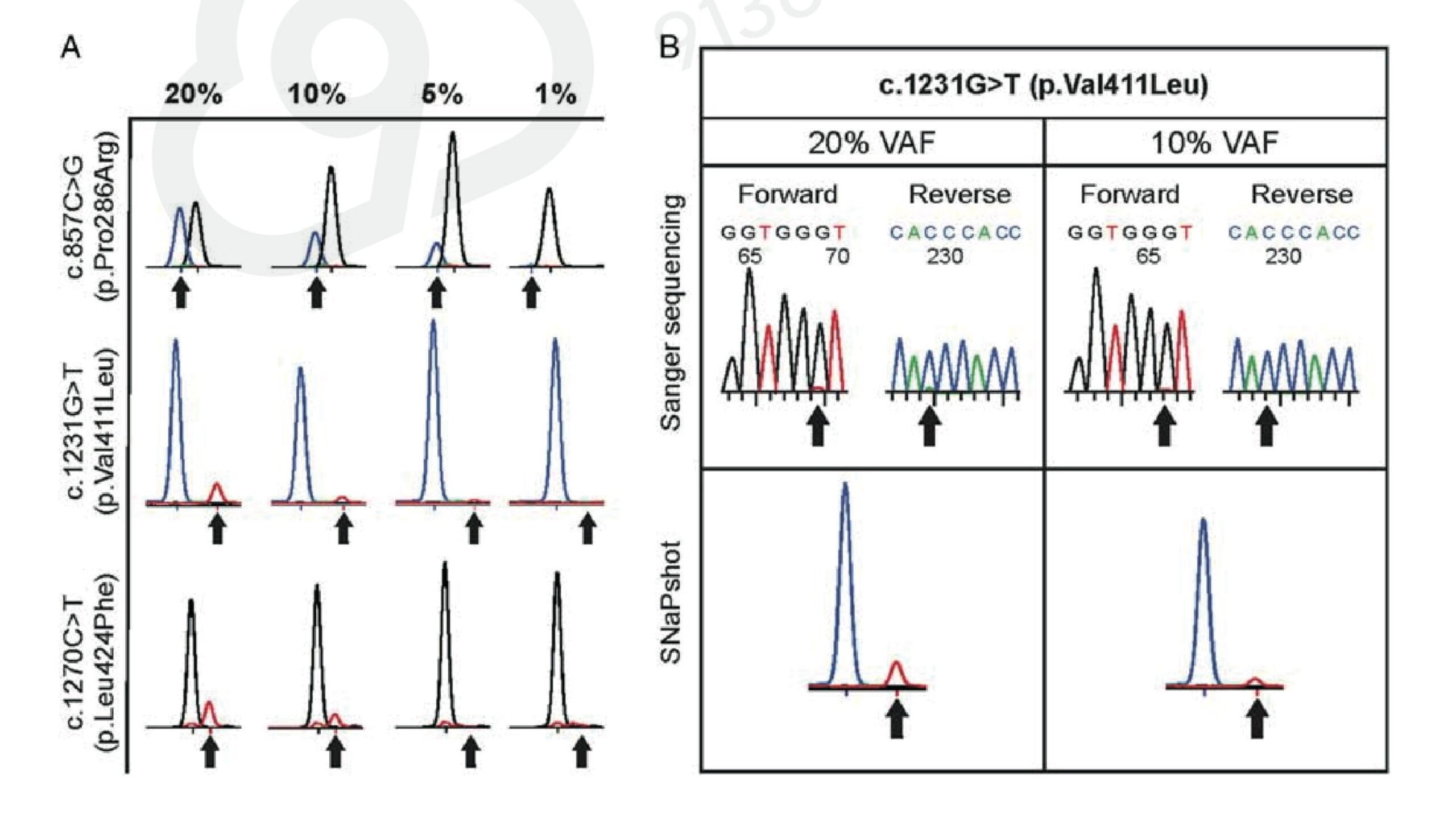
摘要:大多数DNA聚合酶ε(DNA polymerase epsilon,POLE)中核酸外切酶结构域存在致病突变(POLEmut)的“超突变”肿瘤预后良好,即使是高级别组织类型,因此确定子宫内膜癌(endometrial carcinomas,ECs)的POLE突变状态具有重要的临床意义。目前,尚无特异的形态学或免疫表型特征可在不进行分子检测的情况下准确判断是否为POLEmut肿瘤。因此,如果不使用昂贵和/或耗时的DNA测序方法,识别POLEmut肿瘤具有挑战性。我们研发了一种新的SNaPshot检测方法,以促进在ECs中常规且高效的进行POLE突变检测。SNaPshot识别编码POLE核酸外切酶结构域的9、11、13和14外显子中的15个核苷酸变异位点。变异位点的选择依据突变位点的重现性、影响蛋白功能的证据、与高肿瘤突变负荷(TMB)的相关性和/或ECs临床疗效研究的发现。根据文献报道的致病性体细胞变异,预测该方法对ECs的临床灵敏度为90%-95%。在多次的重复试验和倍比稀释试验中,阳性患者(n=11)和阴性对照患者(n=20)均得到预期的基因型结果。验证性研究表明,该方法覆盖变异的特异度和灵敏度均为100%。分析的灵敏度保守估计约为10%的变异等位基因丰度(variant allele fraction,VAF),但也有灵敏度低至5% VAF的报道。正如预期的那样,SNaPshot检测比Sanger测序对低于20%的VAFs更灵敏,这是体细胞变异检测的一个重要特征。我们首次开发和验证了用SNaP-shot检测POLE热点突变。虽然二代测序(next-generation sequencing,NGS)和Sanger测序的方法也被用于检测POLE突变,但在高负荷的病例量背景下,SNaPshot方法为实现分析的灵敏度、成本效益和效率三者之间的平衡提供了可行性。